《医学数据挖掘与实践》实验指导
《医学数据挖掘与实践》实验指导
前 言
通过巩固和加深数据挖掘基本知识的理解,提高运用学习医学数据的数据挖掘,用软件求解操作培养学生的逻辑思维,掌握基本的数据挖掘能力,有利于专业的知识储备。本实验指导共9项实验,分别从R软件的使用、数据预处理、k近邻、决策树、随机森林,聚类、关联规则算法运用医学数据,指导学生操作,实验过程中要求学生能分析实际问题、对学生数据挖掘和分析过程、独立思考等起到指导作用。
实验教材:自编教材
实验学时:32学时
实验一 R与Rstudio安装与基本操作
(综合性实验,2学时)
【实验目的与要求】
1.熟悉R与RStudio软件功能和操作特点。
2.了解RStudio的工具栏和操作方法。 3
3.熟练掌握RStudio环境与基本语法。
4.R包的安装及帮助。
【实验仪器、器材与试剂】
计算机,R与RStudio软件
【实验内容、方法与步骤】
实验内容:
1.安装R和Rstudio
2.打开Rstudio,逐一了解熟悉其中各项功能。
3.Rstudio脚本输入简单的R命令。
4.安装ggplot包
步骤:
1安装R和RStudio
(1)从http://mirror.bjtu.edu.cn/cran/下载R语言相应版本,双击安装;

(2)从https://www.rstudio.com/products/rstudio/download/下载RStudio安装。
说明
(1)R语言本身仅提供了命令行工具,一般用户可能会觉得使用不便,所以用RStudio作为工具来使用R语言;
(2)RStudio仅为R的IDE(集成开发环境),依赖于R;
(3)在苹果系统中,启动RStudio时要求安装command line tools,在弹出窗口中选
择安装即可);
(4)R和RStudio都是免费开放源代码的,所以尽可能在官方网站上下载(R在全球
有很多镜像,等同于官网,上述下载地址即其在北京交通大学的镜像)
2. 运行:
2.1运行R语言
如果不使用RStudio,双击R语言图标即可启动。
启动后如下图所示,在提示符处输入R语言命令
2.2 RStudio启动
RStudio相对于R语言自身提供的命令行工具,使用要便捷得多。
启动后RStudio的环境如下图所示。左侧为同启动R时一样的命行窗口;右上侧为“环境”和“历史”。环境是指当前R语言的运行环境,也称为工作空间,在使用过程中所创建的变量、数据都在这里列出,而用户在使用过程中输入的命令都在“历史”子窗口中列出。
右侧包括“文件”、“绘图”、“扩展包”、“帮助”、“视图”等子窗口。相关功能在后续使用过程中再详细说明。
2.3 退出
在退出时,R和RStudio默认都会弹出窗口询问是否保存工作空间,如果选择保存,则R或RStudio会保存全部的变量、数据等,下次打开R或RStudio时还可以继续使用而
不用再次输入。如下分别为R和RStudio退出时的弹出窗口。
3. R基本语法和计算
R语言运算符号
运算符号:+(加)、-(减)、*(乘)、/(除)、^(乘方)、%/%整除、%%求余; 逻辑判断符号:>(大于)、<(小于)、>=(大于等于)、<=(小于等于)、!=(不等)、 ==(相等)
逻辑运算符号:&(逻辑与)、|(逻辑或,Enter键上边的竖线) 赋值符号:<-或-> 示例: 在命令窗口输入
a<-2
此时,变量a的值就为2。2->a的功能与a<-2一样。赋值符号也可以用=替代,但是在某些情况下会出错,所以不建议在R语言中使用。
完成以下基本计算(将输入和输出一起截图)
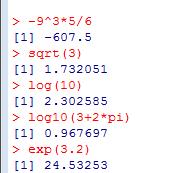
-9的3次方乘以 5再除以 6;(^,*,/)
3 的算术平方根; (sqrt())
10 的自然对数;(log())
以 10 为底的3+2π 的对数;(log10(),pi)
以自然对数为底的3.2 的指数;(exp())
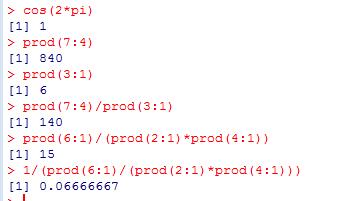
三角函数cos 2π的值;(cos())
连乘计算:①,即7*6*5*4 ;(prod(7:4)) ②3!; ③7*6*5*4/3! ;
组合数计算:① ; ② 1/。(利用上一题)
运行结果截图:
依次输入以下命令,完成以下向量的基本运算,理解体会每个命令或函数的意义(将输入和输出一起截图)
>x<-1:4 #将1,2,3,4四个数赋值给向量x
>a<-10
>x*a
>x+a
>sum(x)
>max(x)
>min(x)
>mean(x)
>median(x) #求x的中位数
>var(x)
>sort(x)
运行结果截图:

4. 安装包获取帮助
>install.packages(“ggplot2”) #安装ggplot2包
>library(ggplot)#加载ggplot包
>help(ggplot) #ggplot解释
>?ggplot #ggplot解释
【实验报告】
安装R和Rstudio,完成实例的基本计算。
实验二 R数据结构和可视化
(设计性实验,4学时)
【实验目的与要求】
1.掌握R中基本数据结构,向量、数组、矩阵、列表、数据框、因子的创建。
2.R数据的读取;掌握数据的基本描述与质量探索
3.ggplot绘制简单的线图,直方图等。
【实验仪器、器材与试剂】
计算机,R与RStudio软件
【实验内容、方法与步骤】
1.创建向量、矩阵、数组、列表、数据框、因子
2.数据的读取。
3.绘制基本图形。
步骤:
1. 1、向量(Atomic Vectors)
向量是用于储存int、character、bool等数据的一维数据结构,单个向量中的数据必须拥有相同类型。使用函数c()来创建向量。R中的向量可以看作是Python中具有相同数据结构的非嵌套列表。
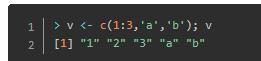
这里尝试在一个向量中存储数值型和字符型数据,可以发现输出结果中所有数据都被转换成了字符型数据。
当不同类型的数据使用c()函数创建atomic vector时,因为atomic vector的元素都必须是同质的,所以系统会依照character, double, integer, logical的优先顺序对数据类型进行转换。
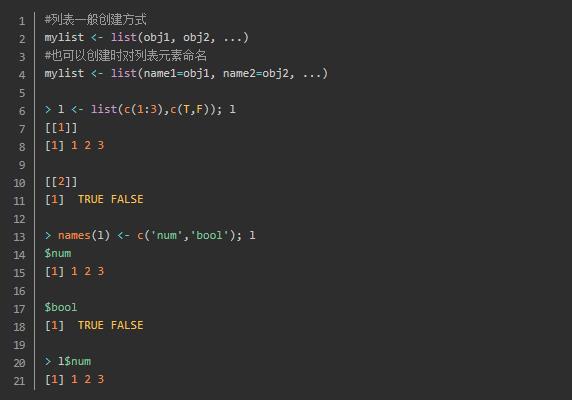
1.2 列表(List)
R中的列表类似于Python中的字典,可以存放各种不同的数据结构和嵌套数据结构,R中列表可以人为的给予其赋予名字(相当于Python字典中的键,也可看做数据框中的列名)未赋予名字时,系统会按索引给R列表中各个数据赋予名字。使用list()创建列表。使用names()可以给列表中的各个元素命名。
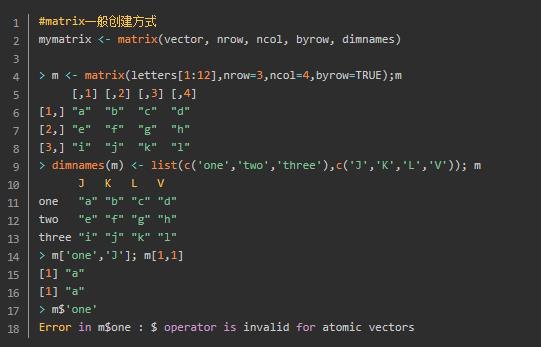
1.3、矩阵(Matrix)
矩阵是一个二维数组,其所有数值类型相同(int,character,bool)。可通过matrix()创建矩阵。matrix可使用下标或行列名来获取元素,但不支持$name的索引方式。可使用dimnames()或rownames()、colnames()对其进行matrix的行列进行重命名。
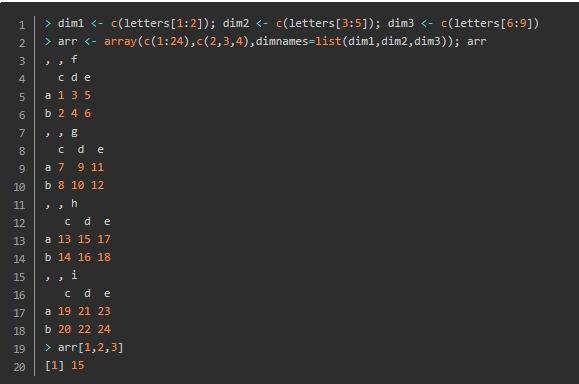
1.4、数组(Array)
数组与矩阵类似,都为同质型的数据结构,但是维度大于2。数组通过array创建。
1.5、数据框(data.frame)
数据框在形式上与矩阵较为类似,但是它每一列的数据类型可以不同,为异质型数据结构。数据框可以使用data.frame()函数来创建,使用colnames()和rownames()来对行列重命名。数据框与python三方库pandas中的DataFrame基本是一样的。
数据框选取元素的方式较为多样,既可以通过下标进行选取,也可以通过列名称来进行选取df$name。同时,在创建数据框时可通过row.names参数指定用来作为行名称的列。
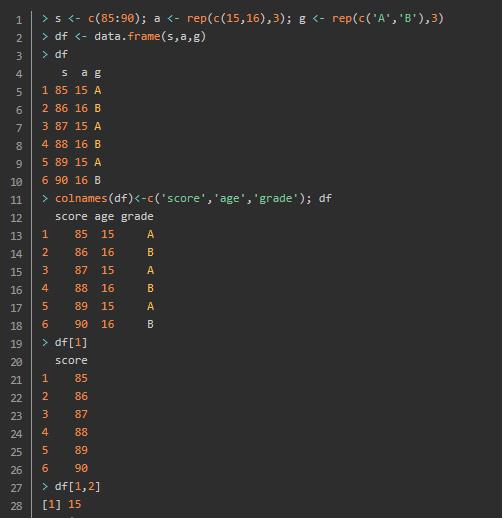
若需要多次选取数据框中的某列数据,可使用attach()、detach()、或with()来简化代码
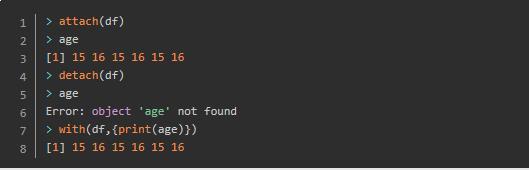
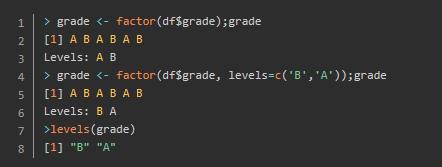
1.6、因子(factors)
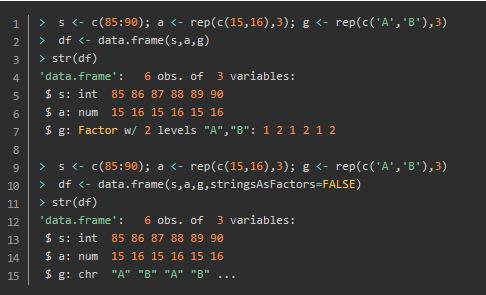
因子比较难以理解,可以将它看作是Python中的set(list),即对向量进行分类计算,每一个唯一值算一个一个分类。他有levels()方法,可以得到一个factor中的所有水平(也即去重后的集合)。他的创建分为三步:(1)将输入的数据转换成character类型;(2)对所有水平进行排序(若未指定排序则自然排序);(3)使用levels中的水平序号重新编码输入的元素。
在构建data.frame时,列元素为character的列经常会自动被转换为factors,这常常不是我们想看到的。通过指定stringsAsFactors=FALSE参数可保留原本的character属性。
2. R数据读取:
在R中进行任何操作和分析工作之前,先需要读取数据。保存在工作目录中的数据可以直接读取,非工作目录的其他位置在读取时需要指明路径。因此第一步工作是了解R的工作目录。下面是具体的代码,输入getwd函数,R返回当前的工作目录。
#查看工作目录
getwd()
[1] "C:/Users/Documents"
你也可以对R的工作目录进行更改,使用setwd函数可以更改工作目录的路径。下面是具体的代码。
#设置工作目录
setwd("C:\\Users\\ r")
设置好工作目录后,开始读取数据,并创建数据表。我们的数据在工作目录下,因此直接读取并命名为loandata。
#读取并创建数据表
loandata=data.frame(read.csv('loan_data.csv',header = 1))
举例:建立一个Excel表,命名为zhihu.xlsx,简单输入几行几列内容如下,sheet名字为nba:
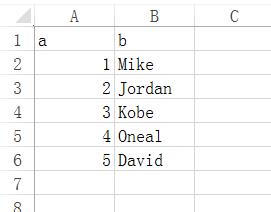
读取方法:使用xlsx包
读取方法1:使用xlsx包

结果显示如下:

3.R绘制基本统计图形,ggplot包绘图
参看书本第二章
【实验报告】
以下是4例病人病历数据,请用在Rstudio中生成名为patientdata的数据框,打印出patientdata,读取糖尿病类型diabetes和病情status这两列;打印出四个病人的年龄信息。 代码使用Rmarkdown完成pdf格式提交,学号姓名实验名称命名。
PatientID | age | diabetes | status |
1 | 25 | Type1 | Poor |
2 | 34 | Type2 | Improved |
3 | 38 | Type1 | Excellent |
4 | 52 | Type1 | Poor |
实验三 数据预处理
(设计性实验,6学时)
【实验目的与要求】
1.掌握tidyverse包相关函数。
2. 掌握dplyr,解决大部分数据处理问题
3.掌握tidyr 清理数据
4.使用readr读入表格数据
5. 熟练应用ggplot2可视化数据法。
【实验仪器、器材与试剂】
计算机,R与RStudio软件
【实验内容、方法与步骤】
1. dplyr数据处理问题
2.使用tidyr 清理数据
3.使用readr读入表格数据
4. 熟练应用ggplot2可视化数据法。
步骤:
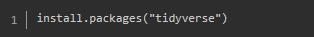
安装tidyverse:
导入:
有用的函数
载入数据
dplyr
过滤
filter()函数可以用来取数据子集。
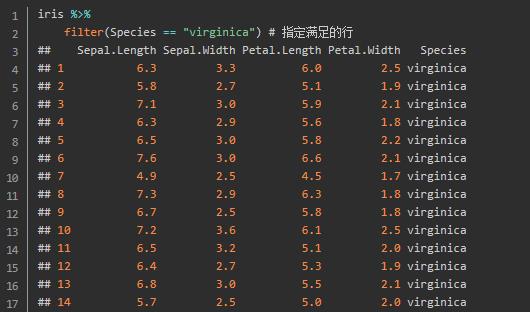
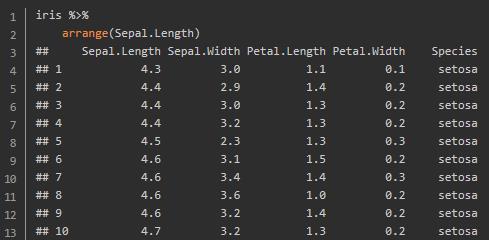
排序
arrange()函数用来对观察值排序,默认是升序。
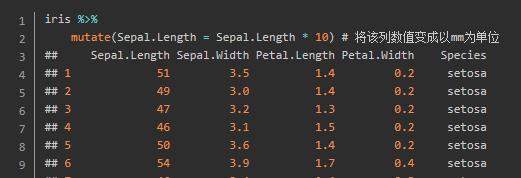
新增变量
mutate()可以更新或者新增数据框一列。
汇总
summarize()函数可以让我们将很多变量汇总为单个的数据点
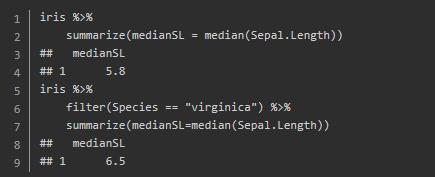
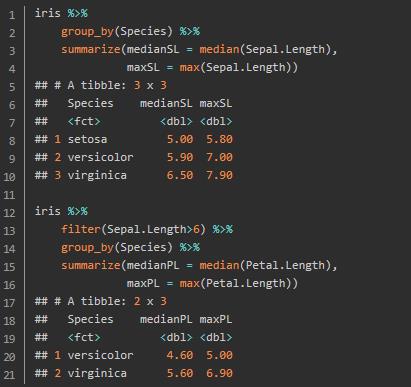
group_by()可以让我们安装指定的组别进行汇总数据,而不是针对整个数据框
ggplot2作图
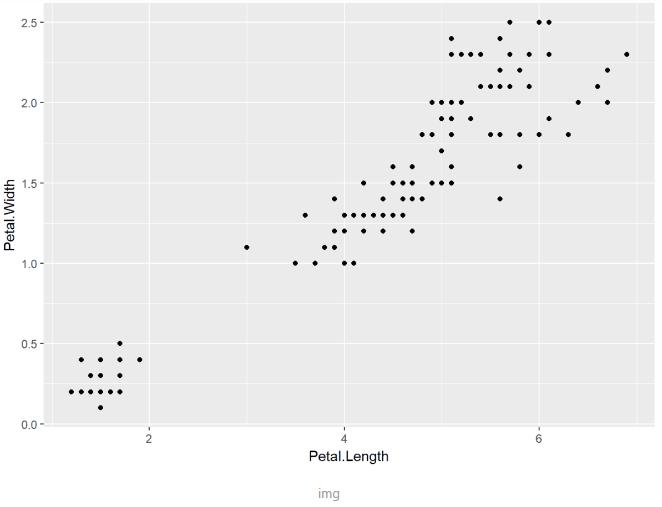
散点图
散点图可以帮助我们理解两个变量的数据关系,使用geom_point()可以绘制散点图:
额外的美学映射
颜色
【实验报告】
用tidyverse包中的gapminder数据集进行数据的预处理。
实验四 k-近邻
(设计性实验,4学时)
【实验目的与要求】
1.加强对k-最近邻算法的理解;
2.锻炼分析问题、解决问题并动手实践的能力。
【实验仪器、器材与试剂】
计算机,R与RStudio软件
【实验内容、方法与步骤】
利用knn预测癌症
肿瘤病人数据集colon.csv,请读取数据集,准备数据,划分训练数据集和测试数据集,建立预测模型,并改进模型预测性能,用KNN算法调整相应K值,提高模型的性能并减少错误分类值的数量,找到最佳预测模型,计算准确率。
代码:
colonData <- read.table("D:/colon.csv",header=TRUE,sep=",")
library(class)
colonKNN <- colonData
set.seed(152)
ind <- sample(2, nrow(colonKNN), replace=TRUE, prob=c(0.8, 0.2))
trainDataKNN <- colonKNN[ind==1,]
testDataKNN <- colonKNN[ind==2,]
train_input <- as.matrix(trainDataKNN[,-2001])
train_output <- as.vector(trainDataKNN[,2001])
test_input <- as.matrix(testDataKNN[,-2001])
prediction <- knn(train_input, test_input, train_output, k=3)
table(prediction, testDataKNN$Class)
【实验报告】
利用 colon.csv的数据进行knn分析,解决程序运行中的问题,使用rmarkdown提交pdf实验报告。
实验五 决策树和随机森林
(设计性实验,6学时)
【实验目的与要求】
1. 掌握决策树和随机森林构建的分类模型;
2.锻炼分析问题、解决问题并动手实践的能力。
【实验仪器、器材与试剂】
计算机,R与RStudio软件
【实验内容、方法与步骤】
数据中的威斯康星州乳腺癌数据集(breast01.txt),数据集变量包括:ID,肿块厚度,细胞大小的均匀性,细胞形状的均匀性,边际附着率,单个上皮细胞大小,裸核,乏味染色体,正常核,有丝分裂,类别。 要求:
(1)读取数据到Rstudio中,将数据类别列转为因子(benign, malignant)并探索数据。
(2)将数据集随机分出训练集和测试集,其中训练集70%,测试集30%.
(3)用rpart()函数创建分类决策树。打印剪枝前后的决策树,输出混淆矩阵,打印模型的准确率。
(4)用随机森林创建预测模型,输出变量的重要性,打印模型的准确率。
(5)比较决策树和随机森林在数据分类的准确率,分类模型选择。
代码如下:
read.table(“breast01.txt)
df <- breast01[,-1]
head(df)
df$class <- factor(df$class,levels=c(2,4), labels=c("benign","malignant"))
(2)set.seed(1234) # 设置随机种子
train <- sample(nrow(df),0.7*nrow(df)) #划分测试数据
head(train)
df.train <- df[train,]
head(df.train)
df.validate <- df[-train,] #划分验证数据
table(df.train$class) #训练集频数统计
table(df.validate$class) #验证数据集频数统计
(3)#使用rpart()创建分类决策树
library(rpart) #加载rpart包
set.seed(1234)
dtree<-rpart(class~.,data = df.train,method = "class",parms = list(split="information")) …(2分)
install.packages("rpart.plot")
library(rpart.plot) #加载rpart作图
rpart.plot(dtree,branch=1,type=1,extra=1, main="Breast Decision Tree1")
dtree$cptable #查看决策树的CP值
plotcp(dtree) #cp值及误差折线图
print(dtree) #查看建立的决策树
summary(dtree) #查看决策树的统计量
dtree.pruned<-prune(dtree,cp=0.0125) #树剪枝
prp(dtree.pruned,type=1,extra=104,fallen.leaves = TRUE,
main="Breast Decision Tree1-pruned") #打印决策树图
dtree.pred<-predict(dtree.pruned,df.validate, type = "class")
dtree.confuse<-table(df.validate$class,dtree.pred) #建立混淆矩阵
dtree.confuse
accuary1<-sum(diag(dtree.confuse))/sum(dtree.confuse) #预测准确度
【实验报告】
利用 breast01.txt的数据建立决策树和随机森林预测模型,比较决策树和随机森林在数据分类的准确率;同时解决程序运行中的问题,使用rmarkdown提交pdf实验报告。
实验六 kmeans聚类
(设计性实验,4学时)
【实验目的与要求】
熟悉R语言的相关对象与函数的用法
掌握利用R进行聚类分析的基本步骤。
【实验仪器、器材与试剂】
计算机,R与RStudio软件
【实验内容、方法与步骤】
依据K-maens函数的使用方法,根据老师给定的数据
1.对数据集预处理,消除量纲差别
2.对预处理后的数据集进行k-means聚类,查看聚类结果,并绘制图形
3.对聚类模型进行优化调优。
iris是R中自带的数据集,读取iris数据集;利用k-means进行聚类分析建模,聚类数为3;使用print函数查看聚类结果;输出混淆矩阵;绘制聚类图并标注每一类的中心点。
rm(list = ls())
mykmeans<-read.csv("Kmeansdata.csv")
kc <- kmeans(mykmeans,3)
print(kc)
fitted(kc)
plot(mykmeans[c("weight", "whr")], col = kc$cluster)
points(kc$centers[,c("weight", "whr")], col = 1:3, pch = 8, cex=2)
【实验报告】
Iris数据集的聚类分析。
实验七 关联规则分析
(设计性实验,6学时)
【实验目的与要求】
熟悉R语言的相关对象与函数的用法
掌握利用R进行聚类分析的基本步骤。
【实验仪器、器材与试剂】
计算机,R与RStudio软件
【实验内容、方法与步骤】
依据关联规则的原理和R中函数使用方法,根据老师给定的数据rules.csv
读取受检者的眼科诊断数据,每个记录包含“眼轴远视”(h52.0),“散光”(h52.2)和“老光”(h52.4)。其中“Y”表示具有该属性,“N”表示不具有该属性。请用arules软件包和arulesViz软件包进行分析,找出其中的关联规则并解释。
1)读取数据文件中的“rules.csv“
2)建立关联规则,设置最小支持度support和置信度confidence分别设为0.1和0.8。
3)查看规则,找出具有较高的置信度和提升度的规则,具有较高的支持度的关联规则。并解释这两条规则的意义。
4)按照提升度对建立的关联规则排序
5)绘制关联规则的散点图和气泡图。
代码:
setwd("C:/Users/15907/Documents/曾学文教学") #设置工作目录
install.packages("arules")
install.packages("arulesViz")
library(arules)
library(arulesViz)
a<-read.csv("rules.csv") #眼轴远视”(h52.0),“散光”(h52.2)和“老光”(h52.4)
rules<- apriori(a,parameter=list(support=0.1,confidence=0.8)) #关联规则运算,支持度support和置信度confidence分别设为0.1和0.8)
inspect(rules)
inspect(sort(rules,by="lift"))
plot(rules, method="graph")
plot(rules, method="grouped",control=list(col=rainbow(5)))
【实验报告】
数据rules.csv进行关联规则分析pdf提交实验报告。
